Forschung
Hochdurchsatzanalyse der Formeigenschaften von Pavement-Zellen
Das Ziel dieses Projektes ist die effiziente Analyse von Formmerkmalen von Zellen. In erster Linie schauen wir uns dabei Pavement-Zellen an, aber die entwickelte Methodik eignet sich prinzipiell für beliebige Zelltypen. Wir haben dazu ein neues Tool zur Hochdurchsatz-Analyse von Pavement-Zellen veröffentlicht, PaCeQuant, das Zellen in Mikroskopbildern automatisch segmentiert und automatisch eine Reihe von Formparametern extrahiert. Im Vergleich zu früher verfügbaren Tools erlaubt der integrierte Segmentierungsansatz dabei, auf einen zeitaufwändigen manuellen Annotationsschritt zu verzichten und größere, repräsentativere Datensätze als bislang üblich zu analysieren.
Publikationen:
- D. Mitra, S. Klemm, P. Kumari, J. Quegwer, B. Möller, Y. Poeschl, P. Pflug, G. Stamm, S. Abel, K. Bürstenbinder, ''Microtubule-associated protein IQ67 DOMAIN5 regulates morphogenesis of leaf pavement cells in Arabidopsis thaliana'', Journal of Experimental Botany, 70(2):529-543, January 2019.
- PaCeQuant: A Tool for High-Throughput Quantification of Pavement Cell Shape Characteristics ,
Kooperationspartner:
- Dr. Katharina Bürstenbinder , Department of Molecular Signal Processing, Leibniz Institute of Plant Biochemistry, Halle (Pressemitteilung des IPB )
- Dr. Yvonne Pöschl , Institute of Computer Science, Martin Luther University Halle-Wittenberg & German Integrative Research Center for Biodiversity (iDiv) Halle-Jena-Leipzig, Leipzig
PaCeQuant ist als Teil von MiToBo und damit als Erweiterung für ImageJ/Fiji implementiert.
- MiToBo website
- Homepage von PaCeQuant
Analyses und Verlaufsprognose von PET-Scans von Hodgkin Lymphome Patienten
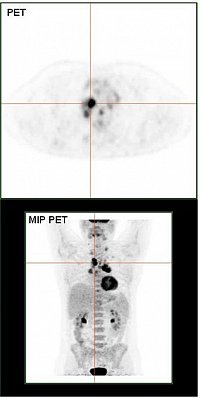
PET Scan (Quelle: https://commons.wikimedia.org/wiki/File:HodgkinLymphomPETCT.jpg, Modifiziert)
Das Hodgkin Symndrom ist eine maligner Tumor des Lymphsystems, dervorwiegend bei Kindern und Jugendlichen auftritt.Die Heilungsaussichten sind mittlerweise sehr gut, jedoch treten insbesonderaufgrund von Radiotherapie starke langfristige Nebenwirkung auf.In diesem Projekt soll auf der Basis von PET-Untersuchungen der Glukoseaufnahme (FDG-PET)prognostischer Faktoren identifiziert werden,die perspektivisch zur Risikobewertung und Therapiesteuerung genutzt werden sollen.Der entscheidene Ansatzpunkt für die Arbeiten ist der aktuelle Stand der Diagnosik,bei dem das Ausbereitungsmuster nur sehr grob berücksichtigt wird.
Das Ausbreitungsmuster des Tumor wird mittles automatischer Segmentierungsverfahrenund geeigneter Merkmale sowohl auf der Ebene von Körperregionen mit hoherGlukoseaufnahme (sowohl Normalgewebe wie auch Tumornester) und auf der Ebenedes gesamten PET scans der Patient*innen charakterisiert.Hierauf aufbauen werden Verfahren des machinellen Lernes genutzt, um Normal- vonTumorgewebe zu differenzieren und eine Verlaufsprognose automatisch zu bestimmen
Kooperationspartner:
Prof. Regine Kluge, Thomas GeorgiKlinik und Poliklinik für Nuklearmedizin,Universität Leipzig
Automatische Segmentierung pflanzlicher Wurzeln in Minirhizotron-Bildern
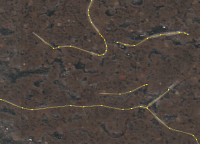
Minirhizotronbild mit Segmentierungsergebnis
Wurzeln sind ein wichtiges Organ von Bodenpflanzen, da sie lebensnotwendige Funktionen erbringen. Hierzu zählen Verankerung im Boden, Ressourcen-Aufnahme, -Transport und -Speicherung. Diese Funktionen stehen im direkten Zusammenhang mit Wurzelbildung und -sterben, deren Verlauf im Rahmen von biologischen Fragestellungen untersucht wird.
Eine zerstörungsfreie Methode der Untersuchung von Wurzeln bietet die Minirhizotrontechnik. Damit können die Wurzeln direkt in der Erde und im zeitlichen Verlauf abgebildet werden. Für die Extraktion der relevanten Daten aus den Bildern, wie Wurzellänge und -dicke, müssen die Wurzeln exakt annotiert werden. Eine manuelle Annotation der Wurzeln ist jedoch eine zeitintensive und mühevolle Aufgabe.
In diesem Projekt soll ein Verfahren zur automatischen Segmentierung der Bilder entwickelt werden, welches eine Detektion der Wurzeln mit weniger (manuellem) Aufwand und in kürzerer Zeit ermöglicht. Hierfür werden verschiedene Ansätze verwendet: Zum einen erfolgt eine Detektion von relevanten Merkmalen, wie Kanten. Zum anderen werden Verfahren des maschinellen Lernens, wie Deep Learning, eingesetzt.
Eine Herausforderung stellen der inhomogene Erdboden und seine Wassereinschlüsse dar. Sie verkomplizieren selbst in der manuellen Annotation die Unterscheidung von Wurzeln und Erde, eine automatische Erkennung der Wurzeln jedoch wird damit erheblich erschwert.
Die Entwicklung eines verlässlichen, automatischen Verfahrens zur Segmentierung von Wurzeln würde es ermöglichen, mehr und größere Datensätze ressourcen-effizient auszuwerten und somit die biologischen Zusammenhänge in Pflanzen noch umfassender zu erforschen.
Kooperationspartner:
PD Dr. Alexandra Weigelt und Dr. Hongmei Chen,
AG Spezielle Botanik und Funktionelle Biodiversität,
Institut für Biologie, Universität Leipzig
rhizoTrak: Eine neuartige Software zur manuellen Annotation von Minirhizotron-Bildern

Mit Hilfe von Minirhizotronen kann Wurzelwachstum zerstörungsfrei beobachtet werden. Dafür werden lichtdurchlässige Röhren in den Erdboden eingebracht und mittels eines Scanners Bilder in den Röhren aufgenommen. Das Verfahren zerstört die Wurzeln nicht, wodurch den Wissenschaftlern die Aufnahme von Bildern im zeitlichen Verlauf erleichtert wird. Dies führt zu einer großen Menge an aufgenommenen Bilddaten. Um diese Daten zu analysieren, müssen sie manuell annotiert werden, womit ein hoher Aufwand verbunden ist. Existierende Annotationssoftware jedoch unterstützt die Annotation von Zeitreihen oftmals nur sehr eingeschränkt.
Aus diesem Grund entwickelt unsere Arbeitsgruppe das Open-Source-Tool "rhizoTrak", welches benutzerfreundlich, effizient und flexibel die manuelle Annotation von komplexen Zeitreihen von Minirhizotron-Bildern unterstützt. rhizoTrak baut auf TrakEM2 auf und erweitert die Software mit für die Annotation von Wurzelbildern speziell benötigten Funktionen. rhizoTrak ist als Fiji-Plugin verfügbar und verwendet offene Datenformate, welche nicht nur für weitere Analysen und statistische Auswertungen genutzt werden können, sondern vor allen Dingen die perfekte Basis für eine Weiterentwicklung und Integration neuer Methoden bieten, wie zum Beispiel zur automatischen Segmentierung.
Weitere Informationen:
rhizoTrak-Webseite, https://prbio-hub.github.io/rhizoTrak/
Biologische und bioinformatische Analyse von TAL-Effektoren in der Interaktion zwischen Xanthomonas oryzae und Reis (DFG-Projekt)

TAL Effektor

RVD-Bindespezifitäten
Projektleiter: Dr. Jan Grau
Dieses Projekt soll unser Verständnis der Interaktion zwischen pflanzenpathogenen Bakterien und ihren pflanzlichen Wirten verbessern. Hierzu werden wir die Funktionsweise von Transcription-activator-like-Effektoren (TALEs) im wirtschaftlich bedeutenden Modellsystem Xanthomonas oryzae und Reis (Oryza sativa) untersuchen. Wir verfolgen einen interdisziplinären Ansatz, der biologische und bioinformatische Arbeitspakete in sich vereint. Im biologischen Teil werden wir TALEs aus mehreren, bisher uncharakterisierten X. oryzae Stämmen identifizieren. Transkriptom-Analysen (RNA-seq) werden zeigen, welche TALE-Zielboxen in-vivo funktionstüchtig sind, und ermöglichen uns außerdem TALE-Zielgene zu identifizieren. Systematische Reporter-Assays von Zielboxen in ihrer Promotor-Umgebung werden es uns erlauben, unterschiedliche biologische Parameter der TALE-Funktion zu unterscheiden. Der bioinformatische Teil wird die Aufschlüsselung der TALE-Repertoires und die Identifikation von Zielgenen unterstützen. Zudem werden die im biologischen Teil generierten Daten in Algorithmen genutzt werden, die weitere Regeln für die in-vivo TALE-DNA-Bindung bestimmen und Eigenschaften von Promotoren oder spezifische Promotorelemente identifizieren, die für die Gen-Aktivierung durch TALEs notwendig sind. Durch diesen ineinandergreifenden Ansatz wird das Projekt maßgeblichen Aufschluss über die biologische Aktivität von TALEs geben, um in Zukunft resistente Reissorten zu entwickeln.
Kooperationspartner:
PD Dr. Jens Boch, Institutsbereich Genetik, Institut für Biologie, Martin-Luther-Universität Halle-Wittenberg
Detektion und Verfolgung subzellulärer Strukturen in Mikroskopbildern
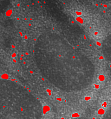
Mikroskopbildaufnahmen von fluoreszierend markierten, subzellulären Strukturen sind ein wichtiges Hilfsmittel in der biomedizinschen Forschung, um Systemprozesse innerhalb einer Zelle und ihre Veränderungen unter äußeren Einflüssen zu verstehen. Die für die Forschung relevanten Strukturen, etwa Stress-Granules und P-Bodies, sind dabei vielfältig und weisen eine große Varianz in ihren Eigenschaften, z.B. in ihrer Größe oder ihrer Homogenität, auf. Entsprechend variabel müssen auch Verfahren zur automatischen Erkennung solcher Strukturen in Mikroskopbildern sein. Wir nutzen dazu unter anderem adaptive Verfahren auf Basis von Wavelets. Zum Tracking von Partikeln in Mikroskopbild-Videos finden ferner Techniken auf Basis probabilistischer Filter Anwendung.
Kooperationspartner:
Sektion Molekulare Zellbiologie, Martin-Luther-Universität Halle-Wittenberg, Prof. Dr. Stefan Hüttelmaier
Zellbildsegmentierung mit aktiven Konturen
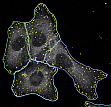
Die große Variabilität in den Eigenschaften und Verhaltensweisen einzelner Zellen innerhalb von großen Zellpopulationen lässt sich in der biomedizischen Forschung nur durch eine Analyse entsprechend umfangreicher Datenmengen erfassen. Dabei sind insbesondere die Unterschiede zwischen einzelnen Zellen von hohem Interesse, so dass qualitative und quantitative Analysen, etwa die Detektion markierter, subzellulärer Strukturen, immer bezogen auf einzelne Zellen erfolgen sollten. Das setzt natürlich eine exakte Lokalisierung der einzelnen Zellen in einer Population voraus. Wir lösen dieses Problem für verschiedenste Zelltypen, unter anderem auch für neuronale Zellen, durch eine Bildsegmentierung mit aktiven Konturen, d.h. sowohl mit Levelsets als auch mit Snakes, und entsprechend adaptierten Energiefunktionen.
Kooperationspartner:
Sektion Molekulare Zellbiologie, Martin-Luther-Universität Halle-Wittenberg, Prof. Dr. Stefan Hüttelmaier
Alida und MiToBo

Die Entwicklung der verschiedenen Algorithmen zur Bildanalyse erfolgt mit dem Ziel einer hohen Modularität und damit Wiederverwendbarkeit. Gleichzeitig sollen sie meistens zeitnah an Anwender weitergereicht werden, so dass neben der reinen Funktionalität auch entsprechende Nutzerschnittstellen geschaffen werden müssen. Um diese Ziele bestmöglich miteinander zu vereinbaren, haben wir ein Konzept entwickelt für eine möglichst integrierte Entwicklung von Algorithmen und Nutzerschnittstellen. Alida (die Kurzform von ''Advanced Library for Integrated Development of Data Analysis Applications'') definiert einen Standard zur Implementierung von Datenanalyse-Algorithmen und ihrer Ausführung, und erlaubt dadurch unter anderem eine automatische Generierung von Benutzerschnittstellen sowie eine automatische Dokumentation ausgeführter Analyseschritte.
Alida wurde inzwischen in Java und prototypisch auch in C++ implementiert. Die Java-Implementierung dient dabei als Basis für MiToBo, ''A Microscope Image Analysis Toolbox'', wo unsere Algorithmen für die Mikroskopbild-Analyse gesammelt werden. Die Toolbox umfasst neben Basisfunktionalität wie Filtern und morphologischen Operationen auch komplexere Ansätze wie aktive Konturen und Wavelet-Segmentierung. Außerdem sind Spezialanwendungen, etwa für die Analyse von Scratch-Assay-Bildern, enthalten. MiToBo baut auf ImageJ auf und bietet eine volle Kompatibilität zu der weit verbreiteten Analyse-Software.
Weitere Infos: http://www.informatik.uni-halle.de/alida bzw. http://www.informatik.uni-halle.de/mitobo

